本地安装与基本配置
本节介绍 Galaxy 的最基本下载安装与使用。最适合的场景为个人电脑,单用户使用的情况。
下载与安装
Galaxy 作为一款开源软件,其代码库托管在 http://bitbucket.org ,先安装 mercurial ,然后用 hg 工具将 galaxy 代码库克隆到本地。
~app$ sudo apt-get install mercurial
~app$ hg clone https://bitbucket.org/galaxy/galaxy-dist/
~app$ cd galaxy-dist
可以用 hg branch 命令查看代码分支是否为 stable,如果是其他分支(galaxy代码库有另一分支'default'),切换到 stable 分支:hg update stable。在生产环境下建议使用 stable 分支的代码。
配置与运行
克隆到本地的 stable 代码,一般Linux系统自带Python就可以直接运行了。
~app$ ./run.sh
运行 run.sh,这个shell脚本程序会自动完成初始化数据,依赖库下载,数据库迁移等一系列操作,当看到终端显示serving on http://127.0.0.1:8080时,可以打开浏览器,访问 http://127.0.0.1:8080 即可看到galaxy的界面。
添加官方 toolshed 中的工具
默认的galaxy只带有基本的工具,对于实际工作中需要的各种分析软件,需要添加到自己建立的 galaxy 实例中。
高通量测序的生物信息学软件大多是基于命令行的开源工具。galaxy利用python语言将这些工具粘合到galaxy实例中,使得用户可以在web界面中直接调用命令行工具对数据进行操作。
galaxy有一个toolshed(工具库)的概念:https://toolshed.g2.bx.psu.edu/(官方维护的toolshed),许多著名的工具已经被移植到toolshed中,可以直接被安装到galaxy里,此外也有许多第三方的toolshed包可以添加,甚至掌握了一些python脚本和galaxy xml规范后,也可以自己添加一些分析工具到galaxy中。
除了分析软件外,toolshed还包含创建的数据类型,以及工作流等。
首先修改配置文件 tool_sheds_conf.xml
~$ cp config/tool_sheds_conf.xml.sample config/tool_sheds_conf.xml
其次在galaxy.ini中配置依赖包的安装目录(上一部分已经添加了这个参数,将依赖包安装在tool_dep),然后添加管理员帐号,比如你之前用[email protected]注册的galaxy实例,就将galaxy.ini中admin_users设置为:
admin_users = [email protected]
重启你的galaxy实例,用[email protected]用户登陆,你就有权限访问http://127.0.0.1:8080/admin,在admin界面可以看到Tool sheds下有Search and browse tool sheds链接,点击后可以看到默认的2个toolsheds源。
进入Galaxy main tool shed,工具列表上访有搜索框,在这里输入你要安装的工具名称比如spades后,回车进行检索。
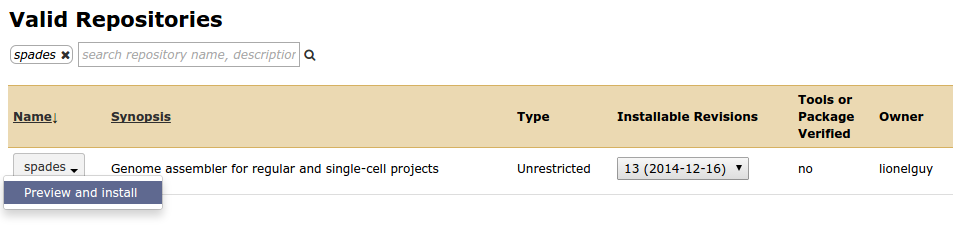
点击结果列表中的spades下拉菜单,选择Preview and install,在转向页面中点击Install to Galaxy后,会出现如下图的提示。
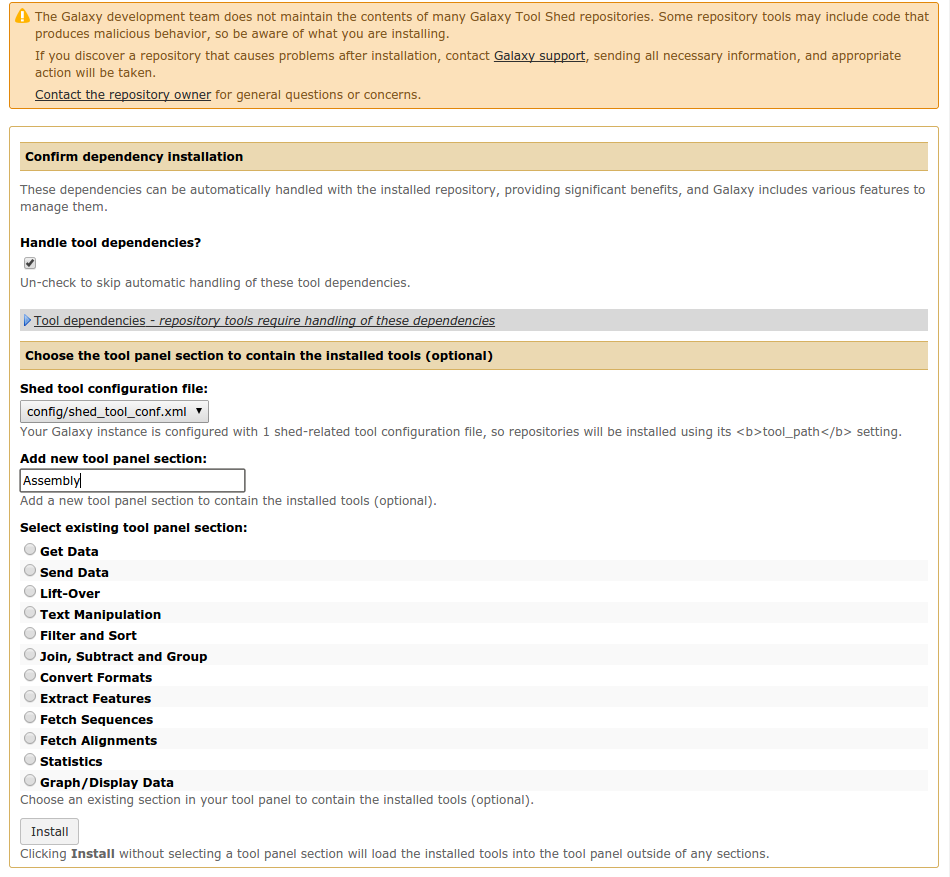
在add new tool panel section中输入Assembly,将spades工具归类到Assembly这个新建的工具类别中,点击页面底部的Install按钮开始安装。