Amazon EC2 教程
Amazon Elastic Compute Cloud (Amazon EC2) 是一种基于 AWS Web 的一项云服务,特点是可在云中提供大小可调的计算量。它的服务接口非常简单,您可以轻松获取和配置容量,可以完全控制您的计算资源,甚至可以直接挂载他人开发的生物信息学工具集合的镜像,减少部署时间。
Amazon EC2 启动新服务器实例的速度非常快,当您的计算要求发生变化时,您便可以快速扩展计算容量。服务按您实际使用的容量和计算量收费,还为开发人员提供了创建故障恢复应用程序以及排除常见故障情况的工具。总之,非常适合作为计算攻击来分析二代测序获得的大数据量分析。
AWS 的新客户可免费试用 Amazon EC2。注册后,新 AWS 客户在一年内每个月都将获得以下 EC2 服务:
- 750 小时运行 Linux、RHEL 或 SLES t2.micro 实例的 EC2 使用时间
- 750 小时运行 Microsoft Windows Server t2.micro 实例的 EC2 使用时间
- 750 小时 Elastic Load Balancing 加上 15 GB 数据处理
- 30 GB 的 Amazon Elastic Block Storage(以任意方式对通用型 (SSD) 或磁性介质型进行组合),附加 200 万次 I/O(采用磁性介质)和 5. 1 GB 快照存储
- 适用于所有 AWS 服务的共计 15 GB 的带宽传出
- 1 GB 区域数据传输
简而言之,新注册用户可以获得1年的有限免费使用。如果要偶尔使用的生产Instance,建议选择按按需实例。这样需要计算的时候打开,计算分析完成后关闭实例即可,可以最大程度上节约资源和开销。对于频繁使用的用户,可以申请专用预留实例,通过支付预付金,可以获得一定的折扣来减低成本。
1. 注册申请
首先要去 https://aws.amazon.com/cn/ 注册一个帐号,帐号申请过程中会要求填写一张信用卡,并会电话联系确认卡的有效性。
用新注册帐号登录后,可以看到 Amazon Web Services 页面,上面列出了 Amazon 所有的服务内容。点击Compute & Networking里的EC2,可以进入你的EC2 Dashboard。
要注意的一点是,顶部工具栏在用户工具右侧有一个地区选项下拉菜单,可以选择不同区域的服务器。个人使用感受,亚洲的服务器比如东京和新加坡的连接速度最快,但是美国的服务器速度也很不错,比一般的VPS商售卖的要快的多!至于选择哪一个地区,可以根据自己的网络实际情况来选择,但是如果你要使用一些AWS Public Datasets,比如Human genome project或者Genbank之类的数据,需要添加volume的snapshot,这些snapshot往往是限定区域的。比如Genbank数据你只能在us-east区域使用,也就是说你要用这个snapshot,你必须把你的Instance建在US East(N.Virginia)区域。
2. 建立 Instance
在EC2 Dashboard左侧列表里点击Instances,然后点击Launch Instance,会弹出如下图所示的一步步配置Instance的界面。
2.1: Choose an Amazon Machine Image (AMI)
在Image界面里选择Ubuntu Server 14.04LTS。

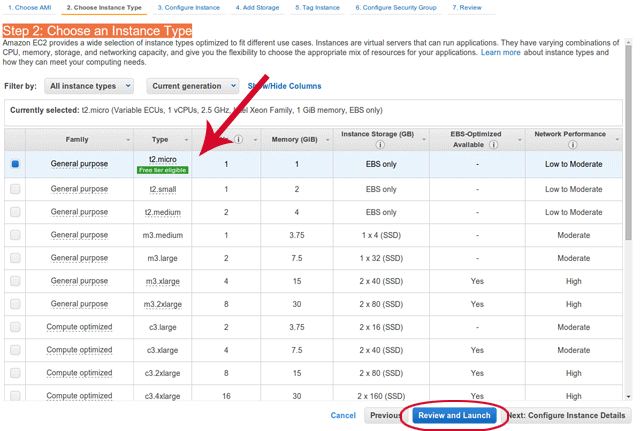
2.2: Choose an Instance Type
Type 选择 t2.micro
是因为这个配置是1年免费的。缺点当然是计算资源比较低,而且当你的计算资源超限额后,会自动把 CPU 性能降低到大概 1/10 左右的情况。要注意的是虽然是免费的,但本质上是1个月720hour的免费计算。如果你同时开了多个 t2.micro Instances,没有在不计算的时候停机stop,仍旧会超出计算时间,被扣美金的。所以如果开了多个Instances,千万别忘了不用的时候关闭。
然后就可以点击review and launch。如果想再定制其他配置可以点击Next: Configure Instance Details。当需要自动加载 docker,或者添加数据Volumes的时候要到后面的步骤里设置。
2.3: Review Instance Launch`
确认配置无误就点击 Launch,会弹出一个Select an existing key pair or create a new key pair选项窗口。这一步很重要,第一次建立新的 Instance 的话,下拉菜单选择Create a new key pair。然后给 key pair 取个好记的名字,比如bac_ngs,然后点击Download Key Pair,就会下载这个密钥文件到你的电脑上。名字叫bac_ngs.pem。这时候底部的Launch Instance按钮就变成可以点击状态,把你的鼠标按上去,你的Instance就开始初始化了。
3. 访问 Instance
如果你的本地电脑操作系统是Linux,可以直接开一个终端程序运行ssh来访问Instance,如果是Windows电脑,推荐使用putty来访问。
3.1 Linux
将下载的 bac-ngs.pem 文件放在一个妥善的位置上,这里放在~/.pem
~/$ mkdir -p .pem
~/$ mv ~/downloads/bac-ngs.pem ~/.pem
~/$ chmod 400 ~/.pem/bac-ngs.pem
现在使用openssh远程访问 AWS Instance 实例。远程的地址可以使用 Dashboard 的 Instance 界面里对应的Public Domain 或者 IP 信息。因为选择的是 ubuntu 操作系统,直接使用 ubuntu 作为用户名,使用 bac-ngs.pem 密钥文件来登录。
~/$ ssh -i ~/.pem/bac-ngs.pem ubuntu@your_public_ip
连接成功的话就能看到 ubuntu 的登录欢迎界面,就可以开始你的生物信息软件配置过程了。
服务器和本地传递数据最方便的方法之一是使用scp命令,如果本地数据要传到服务器的/home/ubuntu/data位置上,可以用下面命令:
~/$ scp -i ~/.pem/bac-ngs.pem ~/data/your_data.fasta ubuntu@your_public_ip:/data
如果是要把服务器上/home/ubuntu/data/001.tar.gz文件传输给本地电脑的话,可以使用以下方式:
~/$ scp ~/.pem/bac-ngs.pem ubuntu@your_public_ip:/data/001.tar.gz .
3.2 Windows
对于 windowsxp 或 windows7 可以使用 putty 来建立与 aws ec2 的 ssh 连接,参考这个教程。windows8 用户可以用 zoc 来访问,具体操作请自行搜索教程。
Running docker in Amazon EC2
docker 是一种虚拟环境,可以很方便的在不同机器上配置环境,近年来快速发展,在云计算服务的领域被高度关注。事实上它的启动速度很快,配置也特别方便,很适合生物信息计算的自动化配置。它对生物学家最大的好处是直接使用他人创建的虚拟镜像,减少安装和部署的学习与工作曲线。或者根据自己的需求自己创建相应镜像,快速部署与使用。
安装 docker
AWS 目前的各个 linux 发行版都已经支持 docker。你可以在上一部分创建好的 AWS EC2 Instance里安装docker:
ubuntu@public_ip:~$ sudo apt-get install docker.io
ubuntu 版本默认的安装包版本比较低,如果想安装最新版本的 docker,需要添加源:
$ sudo apt-key adv --keyserver hkp://keyserver.ubuntu.com:80 --recv-keys 36A1D7869245C8950F966E92D8576A8BA88D21E9
$ sudo bash -c "echo deb https://get.docker.io/ubuntu docker main > /etc/apt/sources.list.d/docker.list"
$ sudo apt-get update
$ sudo apt-get install lxc-docker
运行 docker
接下来我们示例如何安装在docker hub 上他人开发的镜像,假设已经登录到 AWS 并已安装完 docker:
1. 获取镜像
首先我们用 docker 的搜索命令,查找含有名字 ngseasy-fastqc 的所有镜像。你会看到终端会打印出所有含有 ngseasy-fastqc 的镜像。
ubuntu@public_ip:~$ sudo docker search ngseasy-fastqc
因为 docker 镜像非常灵活,可以随时修改他人的镜像成为自己的,所以你旺旺能看到多个不同用户创建的相同镜像,这里我们能看到2个用户 snewhouse 和 compbio 创建的 ngseasy-fastqc 镜像。所以区别不同镜像是通过用户名/镜像名:版本号来区分的。如果不加版本号则已lastest作为默认。
NAME DESCRIPTION
snewhouse/ngseasy-fastqc NGSeasy fastqc
compbio/ngseasy-fastqc
下载镜像 snewhouse/ngseasy-fastqc。
ubuntu@public_ip:~$ sudo docker pull compbio/ngseasy-fastqc
2. 启动容器
启动容器有2种方式,我们需要在容器中进行交互,因此选择基于镜像新建容器并启动。
ubuntu@public_ip:~$ sudo docker run -t -i compbio/ngseasy-fastqc /bin/bash
root@32d858986db7:/# su pipeman
这样你可以开始输入 fastqc 运行程序。
配置适合自己需求的镜像
1. Base image
首先我们需要建立 docker 账号,然后找一个 base image。这里我们用 ubuntu 来创建。首先搜索一下 docker hub 里的 ubuntu 官方镜像。
ubuntu@public_ip:~$ sudo docker search ubuntu | grep Official
2. 创建Dockerfile
docker 镜像在创建容器时,主要通过 Dockfile 来配置,本质上是一个类似 shell 脚本来建立部署的工作流。下面举一个最简单的安装 blast 的 dockerfile 例子。
Dockerfiles
FROM ubuntu:12.04
MAINTAINER My_Name [email protected]
RUN apt-get update && apt-get dist-upgrade -y
RUN apt-get install -y blast
RUN apt-get autoclean && apt-get autoremove -y && rm -rf /var/lib/{apt,dpkg,cache,log}/
更完整的实例可以参考官方文档。
3. 添加工具
ubuntu@public_ip:~$
进一步学习 Docker
如果想对 docker 有更深入的了解,可以先阅读 Docker 从入门到实践,或者阅读 Docker 官方文档。关注 docker 技术的朋友推荐订阅
可用镜像
国外有一些研究者建立了含有不同生物信息分析软件的 docker images,可以根据需要直接下载并运行,避免了在不同机器中部署的各种配置。比如: